This article has multiple issues. Please help improve it or discuss these issues on the talk page. (Learn how and when to remove these messages)
|
| バイオSLAX | |
|---|---|
 | |
 BioSLAX上で動作するさまざまなバイオアプリケーション | |
| 開発者 | シンガポール国立大学 バイオインフォマティクス センター (リソース) Mark De Silva Lim Kuan Siong Tan Tin Wee |
| OSファミリー | Unix系:Linux |
| 作業状態 | 現在 |
| ソースモデル | オープンソース |
| 最新リリース | v 7.5 / 2009年2月5日 (2009-02-05) |
| サポートされているプラットフォーム | IA-32、x86-64 |
| カーネルタイプ | モノリシック |
| ライセンス | 様々な |
| 公式サイト | www.bioslax.com |
BioSLAXは、300以上のバイオインフォマティクスツールとアプリケーションスイートから構成されるLive CD、Live DVD、およびLive USB オペレーティングシステム(OS)です。シンガポール国立大学( NUS)の生命科学研究所(LSI)のバイオインフォマティクスリソースユニットによってリリースされており、CD/DVDまたはユニバーサルシリアルバス(USB)ブートオプションをサポートする任意のPCからブート可能で、Slaxとしても知られるLinux OSの圧縮されたSlackwareフレーバーを実行します。Slaxは、チェコ共和国のTomáš Matějíčekによって、彼自身が開発したLinux Live Scriptsを使用して作成されました。BioSLAXの派生版は、Mark De Silva、Lim Kuan Siong、およびTan Tin Weeによって作成されました。
BioSLAX は、2006 年 4 月に NUS ライフ サイエンス カリキュラムに初めてリリースされました。
歴史
2003年1月、APBioNetはIDRC(カナダ)のPan Asia Networking(PAN)プログラムから研究助成金を受け、APBioGrid構築の一環として、一般的に使用されているバイオインフォマティクスアプリケーションとパッケージをグリッドコンピューティングソフトウェアで統合したAPBioBoxを構築しました。プラットフォームとして選ばれたのは、当時広く普及していたRedhat Linuxでした。同年3月、APBioNetは業界パートナーシップスキーム(AIPS)を立ち上げ、Sun Microsystemsと提携してSolarisプラットフォーム向けのBioBoxを開発しました。6ヶ月後、APBioBoxとSunのbiobox(現在はBio-Cluster Gridと命名)のベータ版が、選ばれた関係者によるベータテスト用にリリースされました。パッケージには、それぞれGlobus Grid Toolkitバージョン2.0とSun Grid Engineが含まれていました。[1]
2003年12月4日、当時APBioBox(Redhat Linux)およびBioCluster Grid(Sun Solaris)と呼ばれていたバイオボックスソフトウェアパッケージは、フィリピン国立研究会議(NRCP)創立70周年を記念して、フィリピン科学技術省(DOST)先端科学技術研究所(ASTI)で開催されたバイオインフォマティクスワークショップでフィールドテストされました。10台のPentiumマシンと数台のSunサーバーがAPBioGridに導入されました。このワークショップとテストされたソフトウェアは、サン・マイクロシステムズが後援し、IDRCが一部資金を提供しました。
2004年7月、NUSのシステム科学研究所(ISS)が主催したワークショップで、Derek Kiong博士はTan Tin Wee准教授に、安定した強力で小型のUnix(Debianベース)プラットフォームとしてKnoppixを紹介しました。2004年9月までに、Ong Guan Sin氏を通じて、NUSの生化学科のLSM2104モジュールの実践コースのプロジェクトとして、APBioBoxのソフトウェアと便利なアプリケーションをプロトタイプであるAPBioKnoppixに組み込むことで、Knoppixリマスターテンプレートを作成することができました。[2]その後、Knoppix 4.02に基づいてアップグレードされ、APBioKnoppix2としてリリースされました。[ 3] APBioKnoppixは広く使用されていましたが、簡単に拡張できないことがわかりました。リマスター前にすべてのアプリケーションが配置されている必要があり、ディストリビューションの柔軟性が非常に低くなっていました。
2005年6月、ライフサイエンス研究所(LSI)バイオインフォマティクスリソースユニットのマーク・デ・シルバ氏は、Slaxのモジュール式システムを採用し、同じベースシステムを使用しながら、アプリケーションファイルや変更点を単一のモジュールに追加することで、様々なツールや変更点を容易にベースシステム上に組み込むことができるという点を評価し、Slaxを新しいバイオベースライブCDのベースとして使用することを提案しました。これにより、Knoppixのように、新しいソフトウェアや変更点が登場するたびにシステム全体を再マスターする必要がなくなりました。
2006 年 4 月までに、BioSLAX の最初のバージョンがいくつかのエディションでリリースされました。
- 標準ユーザーエディション (530 MB)
- 開発者版(700 MB)
- サーバーエディション(470 MB)
BioSLAXはその後、NUSの生命科学カリキュラムにおけるバイオインフォマティクス教育モジュールや、アジア太平洋バイオインフォマティクスネットワーク(APBioNet)傘下のいくつかのイベントで使用されました。APBioNetは、国際計算生物学会(ISCB)の地域支部です。NUSとAPBioNetの両方に対応するために、カスタマイズ版が構築されました。
2007年8月、APBioNetとの協力の下、ベトナム科学技術アカデミーバイオテクノロジー研究所のバイオインフォマティクスリソースサーバーであるBio-IBT(ベトナム・ハノイ)に、カスタマイズされたBioSLAXを用いてベトナムバイオインフォマティクスリソースノードが設置されました。Bio-IBTノードは以下の機能を提供しました。
- BioMirrors 生物学データベースリポジトリ
- NCBI BLASTミラーリソース
- EBI EMBOSSアプリケーションへの Web アクセス
- Clustal W 多重配列アライメントへのウェブアクセス
- T-Coffee多重配列アライメントへのウェブアクセス
- PHYLIP系統推論パッケージへのウェブアクセス
- シーケンス操作スイート、SMS2へのWebアクセス
サーバーに SSH アクセスできるユーザーは、さらに多くのコマンドライン インターフェイスベースのバイオ/ライフ サイエンス アプリケーションにもアクセスできました。
プロジェクト全体は、2007 年 8 月 20 日から 31 日までベトナムで開催された第 1 回 UNESCO-IUBMB-FAOBMB-APBioNet バイオインフォマティクス ワークショップと共同で実施されました。このワークショップは、香港、ハノイ、南沙で 開催された第 6 回国際バイオインフォマティクス会議(InCoB) 2007 のサテライト イベントです。
APBioNet の下で国際機関に導入された BioSLAX の一部のバージョンには、IP を動的に作成された apbionet.org ドメイン名にマッピングできる小さなツールが組み込まれており、これにより各マシンに完全修飾ドメイン名(FQDN) が付与され、インターネット上での存在が実現されました。[引用が必要]
モジュール性
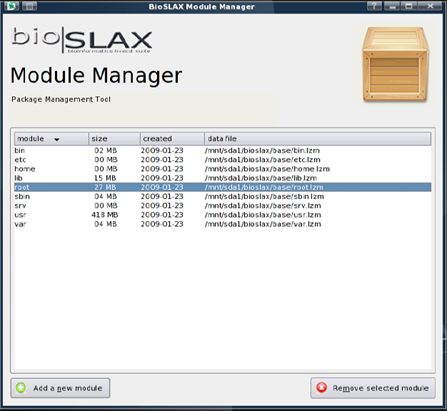
Slaxは「アプリケーションモジュール」をベースLinux OS上にオーバーレイすることで動作するため、ディストリビューション全体がモジュール化されています。システム稼働中でもこれらのモジュールを展開できるという追加機能により、Slaxはさらに魅力的なものとなりました。グラフィカルユーザーインターフェース(GUI)ベースの「BioSLAXモジュールマネージャ」の搭載により、モジュールの動的な追加・削除プロセスが効率化されました。
ユーザーはソフトウェアのアップデートや新バージョンをテストし、必要に応じて以前のバージョンに「ロールバック」することができました。これは、SLAX/BioSLAXがUSBドライブなどの書き込み可能なメディアにインストールされている場合に特に効果的でした。
バージョン
現在までに、BioSLAXにはSlax 5をベースにしたバージョン5.xとSlax 6をベースにしたバージョン7.xの2つのバージョンがあります。バージョン5.xはSlax 5のバージョン番号を踏襲していましたが、バージョン7では、ベースとなっているSlaxのバージョン番号より1つ上の新しいバージョン番号が採用されました。最新バージョンはBioSLAXのウェブサイトからダウンロードできます。[4]
バイオSLAX 5.x
BioSLAX 5.x は主に Slax バージョン 5.1.8 をベースとしており、unionfs を使用した Linux カーネル 2.6 および KDE 3.4 の以前のバージョンを実行していました。
BioSLAX 5.x エディション
標準ユーザーエディション
このエディションはKDE X Window System GUIを実行し、すべてのツールとアプリケーションスイートが含まれていますが、コンパイラツール、Linuxカーネルのソースコード、ヘッダーは含まれていません。主にツールとアプリケーションスイートのみを使用するユーザーに適しています。サイズが小さいためダウンロードしやすく、インターネット帯域幅が限られている地域でも便利です。
開発者版
このエディションはKDE X Window GUIを実行し、すべてのツールとアプリケーションスイート、開発およびコンパイラツールのフルセット、Linuxカーネルのソースコードとヘッダーが含まれています。このエディションは、様々なツールやアプリケーションを必要とし、BioSLAX用の新しいアプリケーションをコンパイルしたり、新しいアプリケーションモジュールを作成したりする必要があるパワーユーザー向けです。
サーバーエディション
このエディションには、X Window GUI、コンパイルツール、Linuxカーネルソース、カーネルヘッダーは含まれていません。主にリモートサーバーとして使用することを想定し、ユーザーはコマンドラインアプリケーションを使用するためにセキュアシェル(SSH)で接続するか、Web経由でサーバーに接続して一般的なバイオアプリケーションへのWebベースポータルにアクセスする必要があります。
NUS LSM版
このエディションは、バイオインフォマティクスの教育のための NUS ライフ サイエンス カリキュラムで使用するためにカスタマイズされた開発者エディションです。
タベルナ版
このエディションは、 TaveRNAを含む開発者向けエディションです。TaveRNAプロジェクトは、ワークフローと分散コンピューティング技術を容易に利用するための言語とソフトウェアツールを提供することを目指しています。
バイオSLAX 7.x
BioSLAX 7.xはSlax 6をベースにしており、Linuxカーネル2.6、KDE 3.5の最新バージョンを搭載し、aufsとlzma圧縮を採用しています。最大の変更点は、クライアントとしてもサーバーとしても使用できることです。また、ディストリビューションがCDからDVDに移行したことで、バージョン5.xでは容量節約のため収録されていなかったアプリケーションが新たに追加されました。Slax 6では、ファイルアロケーションテーブル(FAT)または拡張ファイルシステム(EXT)でフォーマットされたUSBドライブからの起動機能も導入されました。そのため、BioSLAX 7.xにもこの機能が搭載され、CD/DVDでは(再)書き込みができないために利用できない永続的なファイルの処理が可能になりました。
バイオSLAX 8
BioSLAX 7.x以降のバージョンは、ベースディストリビューション(Slax)の開発者であるTomáš Matějíček氏が家族の都合で新バージョンの開発を断念したため、遅延しています。しかし、開発を断念した主な理由は、Squash FSとLZMAがLinuxカーネルにデフォルトで統合され、ユーザーが個別にパッチを適用する必要がなくなるのを待っていたためです。カーネル2.6.38でようやく統合が完了し、Matějíček氏はSlaxの新バージョンを検討することになりました。これにより、今後数ヶ月以内にBioSLAXの新バージョンがリリースされる予定です。Slaxの新バージョンに関する彼の見解は、彼のブログでご覧いただけます。[更新が必要ですか? ]
特徴
標準ツール
BioSLAXは、Linux Slackware 12.1オペレーティングシステムを搭載し、様々なネットワークアダプター用の最新ドライバーを搭載しています。また、様々なワイヤレスカードのサポートも含まれています。さらに、以下のような便利な基本ツールやアプリケーションも多数搭載しています。
- Perl(BioPerlモジュールを含む)
- PHP
- アパッチII
- MySQL
- オープンオフィス
- KPDFリーダー
- モジラファイアフォックス
- モジラサンダーバード
- gFTP
- プロFTPd
- オープンSSH
- Kopeteインスタントメッセンジャー
- VNCビューア
- リモートデスクトップサービス
バイオインフォマティクスツール
バイオインフォマティクスのツールとアプリケーションは、主に 3 つのカテゴリに分類されます。
コンソールアプリ
- ブラスト
- ブラストCL3
- バイオグレップ
- クラスタルW
- エンボス
- ジェネスプライサー
- グリマーHMM
- ハマー
- モデラー
- パムル
- フィリップ
- プライマー3
- R(プログラミング言語)とBioconductor
- Tコーヒー
デスクトップアプリ
- 活動
- アルテミス
- ClustalX (GUI ベースのClustal W)
- Jアライナー
- ジャルビュー
- jEMBOSS (Java EMBOSSスイート)
- ジェイモル
- NJPlot
- ピモル
- シーケンスを読む
- ツリービュー
- Weka(機械学習)
ウェブアプリ
ハードディスクにインストール
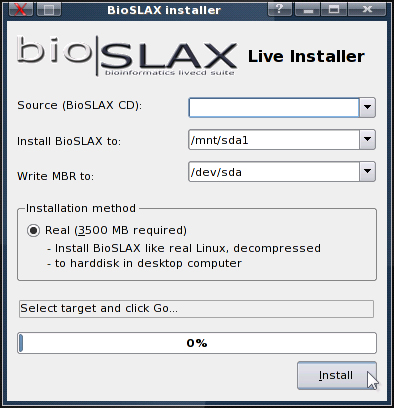
Slax ベースのディストリビューションの便利な点は、ライブ OS を、およそ 3.5 GB のスペースを使用する任意の PC のハード ドライブにインストールされた完全な Linux システムに変換するのがいかに簡単かということです。
KDE Kommanderツールキットで作成されたBioSLAXインストーラーというツールは、ライブOSを完全なLinuxインストールに簡単に変換するために提供されています。モジュールを使用してディストリビューションをカスタマイズし、インストーラーを使用することで、ユーザーは完全にインストールされたカスタマイズされたクライアントを迅速に導入できます。
今後の計画
BioSLAXのアップデート
BioSLAXは、Slackware(またはSlax)の新しいバージョンがリリースされるたびにアップデートされます。ツールとアプリケーションスイートも重要な変更がないか監視され、必要に応じてアップグレードされます。一部のツールは、同じ機能を備えながらも機能が追加され、効率性が向上した他のツールを導入するため、削除される可能性があります。ReadSeq、Primer3、GenesplicerなどのWebベースのポータルも検討中です。
グリッド展開
開発者たちは、様々なグリッドコンピューティングプラットフォームをBioSLAXに統合することも検討していました。BioSLAXはあらゆるCD/DVD/USBからすぐに起動できるため、迅速に導入可能なグリッド対応オペレーティングシステムとして利用できます。そのようなグリッドプラットフォームの一つがUniva Gridプラットフォームです。Univa Grid MPエージェントは、 GridAsia 2009のTan Tin Wee氏による講演で、BioSLAX上でモジュール化されたエージェントを使用することで、任意の場所にあるマシンを別の場所にあるマスターノードのスレーブノードとしてグリッド対応にすることができ、実質的に「グローバル規模のグリッド」を構築できることが示されました。
クラウド上のBioSLAX
開発者たちは概念実証の取り組みとして、VMWareの ESXiとCitrix Xenのハイパーバイザーの両方を使用して、リソースプール上にBioSLAXをインスタンスとして展開することに成功しました。彼らの目標は、学生やスタッフが研究・教育のために任意の数のBioSLAXサーバーを動的にインスタンス化できる「BioSLAXクラウド」を効果的に構築すること(学生がX-Win32、VNC、Exceed、NoMachine NXなどの適切なX Windowクライアントを介してサーバーに接続することで、バイオインフォマティクスの実習を行う)か、UD Grid MPAgentと組み合わせて使用することで大規模ジョブを処理するためのクラスターを形成できるような方法で展開することでした。
この概念実証は、NUSの生命科学カリキュラムの研究・教育に導入され、大きな成功を収めました。2011年には、VMWareのvSphereサーバーとCitrix Xenサーバー上で稼働するBioSLAXクラウドインスタンスが、APBioNetプロジェクト「BioDB100」で多数使用されました。バックエンドの制御と自動化は、Mark De Silva氏によってvSphereとXenの各種APIを用いて作成・実装されました。
開発者たちは2009年から2010年にかけてAmazonとも協議を重ね、Amazon EC2上に同様のBioSLAXクラウドイメージを展開し、研究・教育用マシンの一部をAmazonに移行することでハードウェアコストの削減を目指していた。しかし、BioSLAXイメージをクラウド上で動作させるために必要な完全なハードウェア仮想化をAmazonがサポートしないことが明らかになったため、協議は決裂した。Citrix Xenハイパーバイザーを使用する商用クラウドプロバイダーの大半は、準仮想化のみをサポートする方針だ。これらの事業者の考え方が変わるまでは、完全なハードウェア仮想化が設定されたCitrix Xenハイパーバイザーで動作するプライベートクラウド、またはVMWare vSphereクラウドだけがBioSLAXを実行できるクラウドとなるだろう。
参照
参考文献
- ^ 「アジア太平洋バイオグリッドイニシアチブ」。
- ^ 「APBioKnoppix」。
- ^ 「APBioKnoppix2」。
- ^ “BioSLAX - BioInformatics LiveCD Suite”. 2016年10月3日時点のオリジナルよりアーカイブ。
外部リンク
- 公式サイト
- シンガポール国立大学
- シンガポール国立大学バイオインフォマティクスセンター 2005年8月26日アーカイブ - Wayback Machine
- シンガポール国立大学生命科学研究所
- アジア太平洋バイオインフォマティクスネットワーク
- BioDB100プロジェクトは2011年6月11日にWayback Machineにアーカイブされました
- ユニバ

